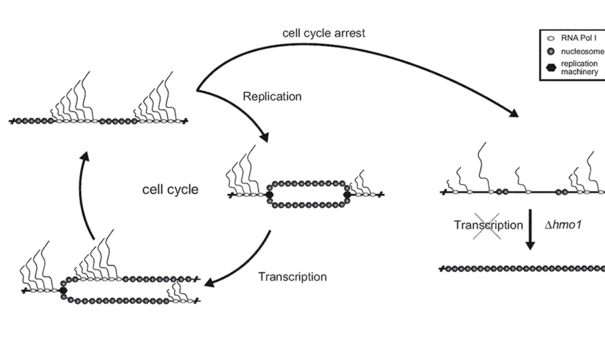
Rekodierungsmechanismen in Infektionen
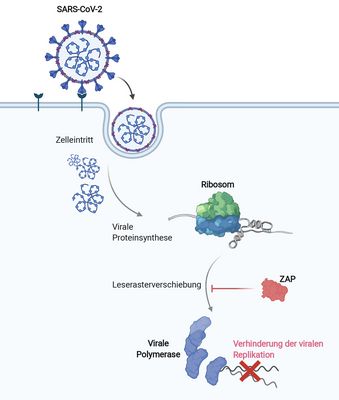
In Viren oder zellulären Genen bestimmte RNAs können während der Translation auf alternative Weise gelesen werden, was als Rekodierung bezeichnet wird. Es ist jedoch unklar, wie genau die Rekodierung durch den Wirts oder virale Faktoren reguliert wird. Ein genaues Verständnis der Rekodierung und ihrer Regulation kann daher der Schlüssel zur Entwicklung neuer RNA-basierter Therapien im Kampf gegen Infektionen sein.Unser Team hat mehrere virale RNA-Interaktionspartner identifiziert, von denen wir gezeigt haben, dass sie die Synthese des SARS-CoV-2-Polyproteins oder HIV-1 replikation stört. Darauf aufbauend untersucht meine Gruppe, wie diese Proteine während der Translation dynamisch mit strukturierten RNAs und dem Translationsapparat des Wirts interagieren. Wir arbeiten derzeit auch an der Identifizierung peptidebasierender oder synthetischer Moleküle, die spezifisch mit viraler-RNA-Elementen interagieren können, da die Modulation der RNA eine wirksame antivirale Strategie darstellt.
Regulation von Translation in Infektionen
Veränderungen in der Umwelt, zelluläre Differenzierung und Infektionen können die Mechanismen der Genexpression beeinflussen und dadurch das zelluläre Proteom auf unerwartete Weise verändern. Dennoch ist es häufig unklar, inwieweit menschliche Zellen nicht standardisierte Translationsereignisse, beispielsweise während einer Infektion, tatsächlich nutzen.
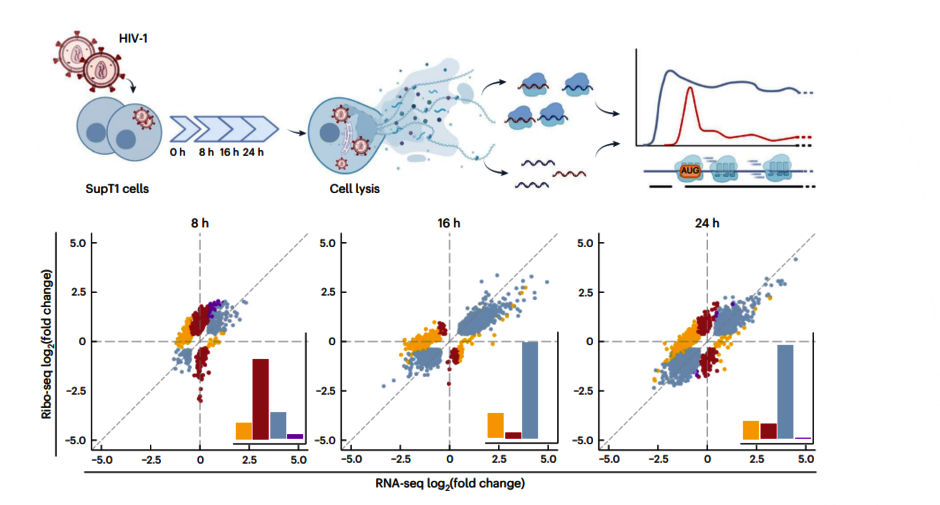
Um die Translationprozesse und die Rolle der RNA-Moleküle in diesen Prozessen besser zu verstehen, setzen wir modernste RNA-Analytik wie Ribosomen-Profiling und Tiefensequenzierung ein. Unser Fokus liegt auf der Dynamik nicht standardisierter Translationsmechanismen sowie der potenziellen Rolle zellulärer Faktoren in diesem Zusammenspiel. Letztlich möchten wir die Interaktion zwischen der Genexpression des Wirts und des Pathogens umfassender entschlüsseln.
Einzelmolekülanalyse von RNA Komplexen
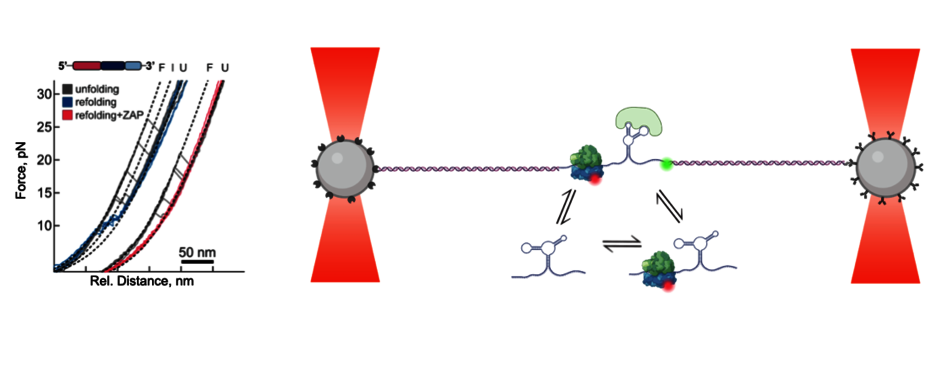
RNAs können in verschiedenen Formen existieren und mit anderen regulatorischen Elementen wie ncRNAs, kleinen Molekülen und Proteinen interagieren, um die Bedeutung der in der Primärsequenz der mRNA kodierten Botschaft zu verändern. Wie RNA-Strukturen und regulatorische Elemente alternative Translationsereignisse steuern, ist derzeit noch nicht vollständig verstanden. Eine Schlüsselfrage, mit der wir uns befassen, lautet: "Inwieweit definiert die Stärke der RNA-Basenpaarungs-Interaktionen und die Konformationsdynamik der Struktur die Neigung der Ribosomen, sich in ein alternatives Leseraster zu bewegen?" Mit Hilfe modernster Einzelmolekül- und Ensembleanalyse-Tools untersuchen wir, wie trans-wirkende Faktoren die RNA-Struktur verändern. Die von uns entwickelten Werkzeuge dienen als Einstiegspunkt für die Entwicklung potenter und spezifischer Modulatoren des Frameshifting.